Duran Clélia

- Équipe de rattachement : BIOFEEL
- clelia.duran*at*univ-pau.fr
Thèse de doctorat (2020-2023)
Ecole doctorale de l’Université de Pau et des Pays de l’Adour (UPPA) : ED 211 SEA
Formation d’origine :
Master Chimie et science du vivant : Biologie moléculaire et Microbiologie Environnementale à l’UPPA 2017-2019
(2020-2023) Outils Moléculaire pour évaluer le fonctionnement des écosystèmes côtiers
Mots clés : Bioindicateur, Ecologie microbienne, Environnement, Outils moléculaires
Responsables scientifiques :
![]() Cristiana Cravo Laureau, Maitre de conférences, Université de Pau et des Pays de l’Adour, IPREM UMR5254
Cristiana Cravo Laureau, Maitre de conférences, Université de Pau et des Pays de l’Adour, IPREM UMR5254
![]() Christine Dupuy, Professeure première classe, Université de La Rochelle, LIENSs UMR7266
Christine Dupuy, Professeure première classe, Université de La Rochelle, LIENSs UMR7266
![]() Hélène Agogué, Chercheure CNRS, Université de La Rochelle, LIENSs UMR 7266
Hélène Agogué, Chercheure CNRS, Université de La Rochelle, LIENSs UMR 7266
![]() Robert Duran, Professeur Classe exceptionnelle, Université de Pau et des Pays de l’Adour, IPREM UMR5254
Robert Duran, Professeur Classe exceptionnelle, Université de Pau et des Pays de l’Adour, IPREM UMR5254
Financement : Allocation de la région Nouvelle Aquitaine
Outils Moléculaire pour évaluer le fonctionnement des écosystèmes côtiers
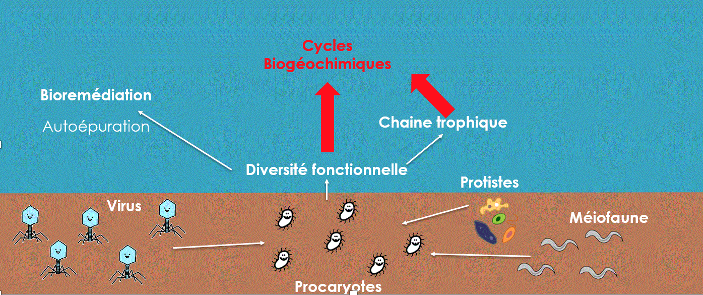
De plus, elles ont démontré des capacités de bioremédiation dont le fonctionnement reste cependant peu connu. Il s’agit de communautés dont la régulation est complexe, contrôlées à la fois par des facteurs biotiques et abiotiques. Entrent en jeu notamment les interactions trophiques avec le reste de la communauté benthique, tel que la méiofaune et la microfaune (protistes) mais également un contrôle des populations par les activités virales (bactériophages et virus d’archées). Les écosystèmes benthiques sont donc d’une importance primordiale pour le fonctionnement des environnements côtiers. Il n’existe cependant pas de bioindicateur moléculaire rendant compte de l’état écologique des sédiments côtiers à l’heure actuelle. L’objectif de cette thèse est de parvenir à mettre en place un tel outil, prenant en compte la diversité trophique des environnements benthiques.
Les objectifs spécifiques de la thèse sont : i/ développer des outils moléculaires ciblant la communauté benthique, allant des virus de bactéries et archées jusqu’à la méiofaune, et incluant les procaryotes et eucaryotes unicellulaires, ii/ mettre en évidence les interactions biotiques, notamment les interaction trophiques, iii/ comprendre les facteurs contrôlant les associations microbiennes dans les sédiments côtiers.
Afin de détecter les divers organismes de la communauté benthique, il est nécessaire de développer des amorces permettant d’amplifier et de séquencer des gènes des communautés virales, protistes et de la méiofaune. Pour cela, deux approches ont été choisies : i/ analyse in silico et basée sur la littérature ; ii/ analyse du métagénome d’échantillons environnementaux. De plus, une communauté artificielle (mock) représentative des milieux benthiques (eau douce, salée et saumâtre) a été mise en place afin de valider d’une part, un protocole d’extraction d’ADN, notamment pour la méiofaune, et d’autre part, les amorces qui seront choisies.
Plusieurs campagnes d’échantillonnage ont été réalisées sur des sites salés, saumâtres et doux afin de mieux appréhender le fonctionnement des écosystèmes benthiques et d’éventuelles variations liées à la salinité. Leur étude permettra d’appréhender le fonctionnement des écosystèmes benthiques en fonction des éventuelles variations de salinités. Un suivi temporel de certains sites permettra également d’évaluer les changements saisonniers.
Les travaux actuels se focalisent sur le design d’amorces ciblant les virus (de bactéries et d’archées) et les eucaryotes. À terme, ces outils permettront d’analyser les communautés benthiques et mettre en évidence les interactions biotiques. À court terme, la priorité est mise : i/ sur les analyses métagénomique afin d’identifier les virus et organismes les plus abondants, ii/ valider le protocole d’extraction d’ADN et les amorces sur la « mock ». Les résultats attendus feront l’objet d’un article, iii/ réaliser les analyses et extractions d’ADN des échantillons (200) des différentes campagnes de terrain. À plus long terme, les amorces seront utilisées pour analyser les échantillons environnementaux via une approche de barcoding afin de décrire les assemblages des communautés benthiques. Ces informations permettront de définir des bioindicateurs.
Activités d’enseignement :
- TD d’Immunologie aux L3 Sciences de la Vie à niversité de Pau et des Pays de l’Adour.